
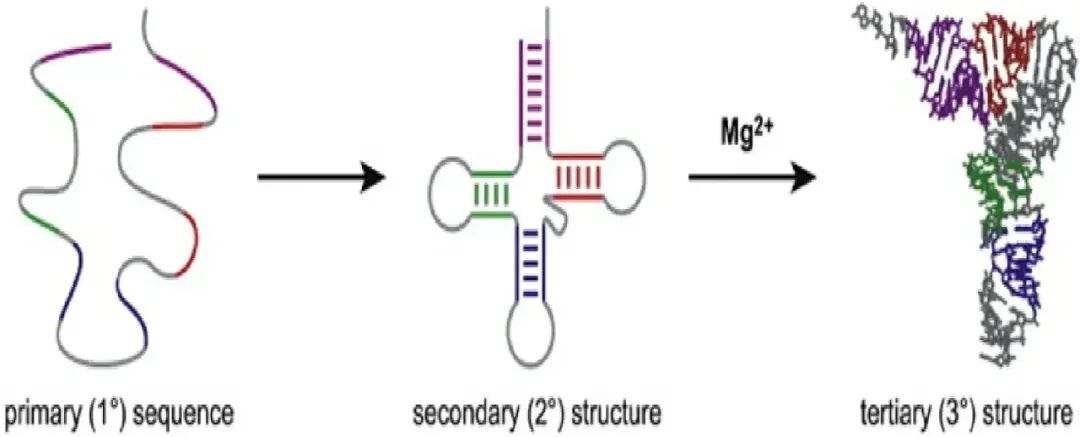
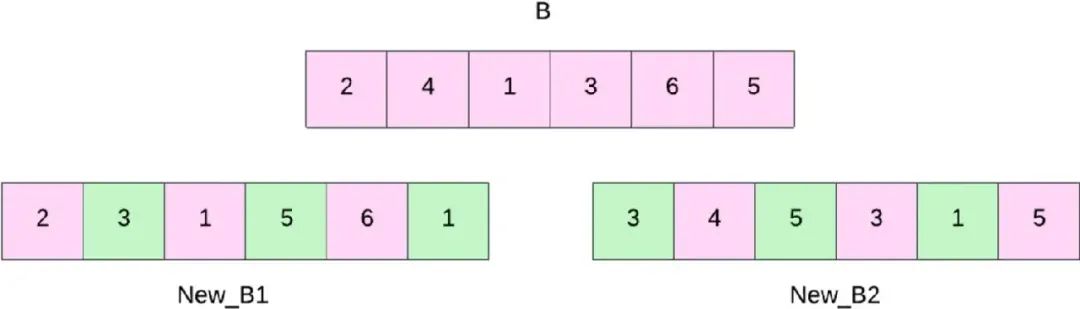
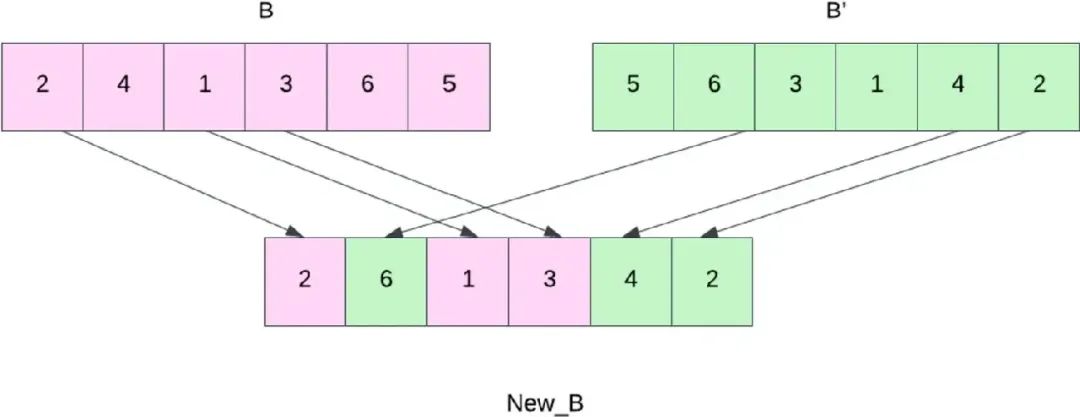
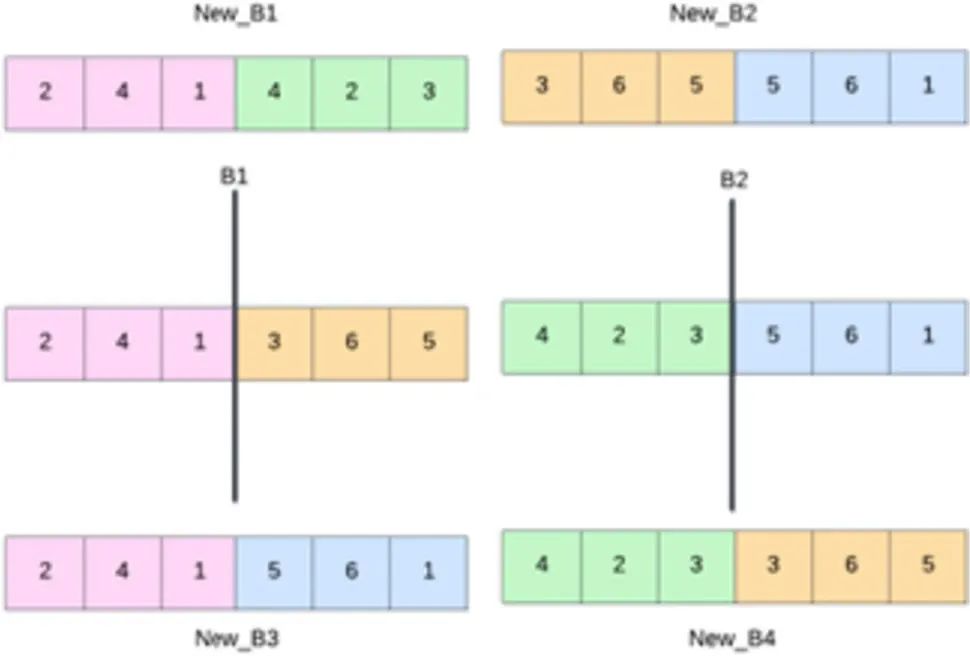
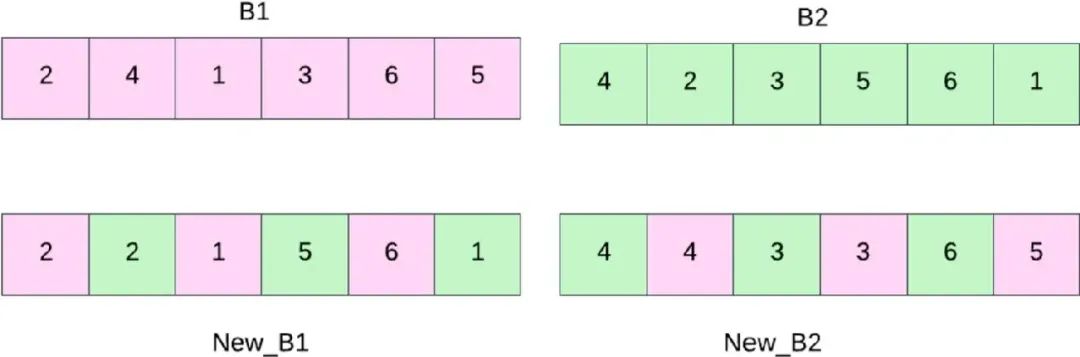
近日,一篇由孟加拉国库尔纳大学(Khulna University)和英国东伦敦大学(University of East London)学者合作的论文“Prediction of RNA Secondary Structure Using Butterfly Optimization Algorithm”(译:利用蝴蝶优化算法预测RNA二级结构)在国际期刊《以人为中心的智能系统(英文)》(Human-Centric Intelligent Systems, HCIN, eISSN: 2667-1336)上公开发表(DOI:https://doi.org/10.1007/s44230-024-00062-6)。 一句话概要: 基于群体的元启发式蝴蝶优化算法预测RNA二级结构,助力新药发现及遗传病研究。 研究动机: RNA在蛋白质合成、DNA 复制、基因表达、细胞分化和遗传进化中具有重要意义。如图1所示, RNA中存在着三级结构。一级结构是 由通过磷酸二酯键连接的核苷酸线性序列。二级结构(二维碱基对)是由同一单链RNA上核苷酸的互补碱基对形成。三级结构指RNA分子在二级结构的基础上进一步折叠形成的复杂三维结构,其结构特点表明,RNA 的二级结构不仅是理解其三维结构和功能的关键桥梁,还能够揭示 RNA 分子的进化特征和相互作用机制。因此,为了深入研究遗传性疾病的病理并辅助生物学家发现新的治疗方法,研究人员一直致力于确定 RNA 的二级结构,从而分析细胞中各类物质的重要性。尽管物理分析方法如核磁共振(NMR)和X射线晶体学在预测RNA结构方面取得了一定的进展,但这些方法操作复杂、耗时且成本高昂。此外,RNA 二级结构预测问题属于NP(Nondeterministic Polynomial time-Hard Problem)困难问题,会造成预测算法准确率随着RNA序列中分子的增加而降低,极大的限制了算法的性能。 ▲ 图1 RNA的三种折叠结构。 研究方法与发现 针对上述问题,本文提出一种基于群体的元启发式蝴蝶优化算法(Butterfly Optimization Algorithm,BOA),用于预测RNA的二级结构。该算法受蝴蝶探索食物现象启发,以蝴蝶对香味的敏感性为前提进行局部和全局搜索,从而找到稳定结构和最佳搜索方案。 具体而言,首先设计了四个搜索算子进行分离全局搜索、反向全局搜索、交换局部搜索和边缘局部搜索。分离全局搜索(图2)将每个分子或结构分成两个部分,注入有助于找到全局最小值点的随机元素。反向全局搜索(图3)结合两个不同区域的局部极小点,搜索全局最小结构。交换局部搜索(图4)在搜索空间的局部区域中交换不同的单体位置,并且像变异算子一样工作,在分子之间产生微小的变化。边缘局部搜索(图5)在局部区域内选择两个不同的结构,并对这两个结构之间的奇偶位置进行边际搜索,生成局部搜索极小点。此外,“优化”功能会丢弃结构中重复编号(如图2中New_B1 重复编号1),从而加快搜索过程。 在RNA STRAND v2.0数据集上,从TP、FP、FN、Sensitivity、Specificity、F-measure和INF等评价角度,与目前主流算法SA、GA、COIN、TL-PSO、FOA和CRO等进行了对比实验,结果表明,所提出的BOA算法在预测RNA二级结构上均优于当前最先进的算法。 ▲ 图2 分离全局搜索。 ▲ 图3 反向全局搜索。 ▲ 图4 交换局部搜索。 ▲ 图5 边缘局部搜索。 研究贡献: 1)提出的蝴蝶优化算法BOA设计了分离全局搜索、反向全局搜索、交换局部搜索和边缘局部搜索,可实现同时进行局部和全局搜索,具有更低的全局搜索过程的时间复杂度; 2)引入热力学模型来选择具有最小吉布斯自由能的稳定二级结构,进一步提高了预测效率。 关于期刊 Human-Centric Intelligent Systems(eISSN:2667-1336)是一本国际化的,经过严格同行评审的开放获取期刊,致力于传播 “以人为中心的智能系统” 中所有相关理论和实际应用的最新研究成果,并提供以人为中心的计算与分析领域的前沿理论和算法见解。为了鼓励科研成果的传播,本刊暂不收取文章处理费。
























 上一篇
上一篇










